CRISPR/Cas9 đang thu hút sự quan tâm của hầu hết các nhà khoa học trên toàn thế giới trong lĩnh vực chỉnh sửa bộ gen và các lĩnh vực sinh học khác cũng được hưởng lợi từ kỹ thuật này. Được khám phá từ một cơ chế tự vệ trong vi khuẩn, hệ thống này có thể dễ dàng lập trình để cắt và chỉnh sửa bất kỳ trình tự DNA nào ở các loài khác nhau. Vậy, cơ chế của nó như thế nào? Ứng dụng vào những gì? Quy trình thực hiện ra sao? Liệu ngoài những ưu điểm, CRISPR/Cas9 có nhược điểm nào không?
I: Cơ chế hoạt động của hệ thống CRISPR/Cas9
Bước quan trọng đầu tiên trong việc chỉnh sửa bộ gen của một sinh vật là lựa chọn trình tự gen mục tiêu. Trong hệ thống CRISPR/Cas9 bao gồm hai đại phân tử chính bao gồm một protein Cas9- một protein có hoạt tính như một enzyme cắt tại vị trí chuyên biệt (endonuclease) và một RNA dẫn đường (guide RNA), tương tác nhau để tạo thành một phức hợp có thể xác định trình tự mục tiêu với độ chọn lọc cao.
Các protein Cas9 chịu trách nhiệm cho việc định vị và cắt DNA mục tiêu, cả trong tự nhiên và trong các hệ thống CRISPR/Cas9 nhân tạo. Một protein Cas9 có cấu tạo gồm 6 tiểu phần: REC I, REC II, cầu nối Helix, PAM Interacting, HNH và RuvC (Hình 1) (Jinek et al. 2014; Nishimasu et al. 2014)
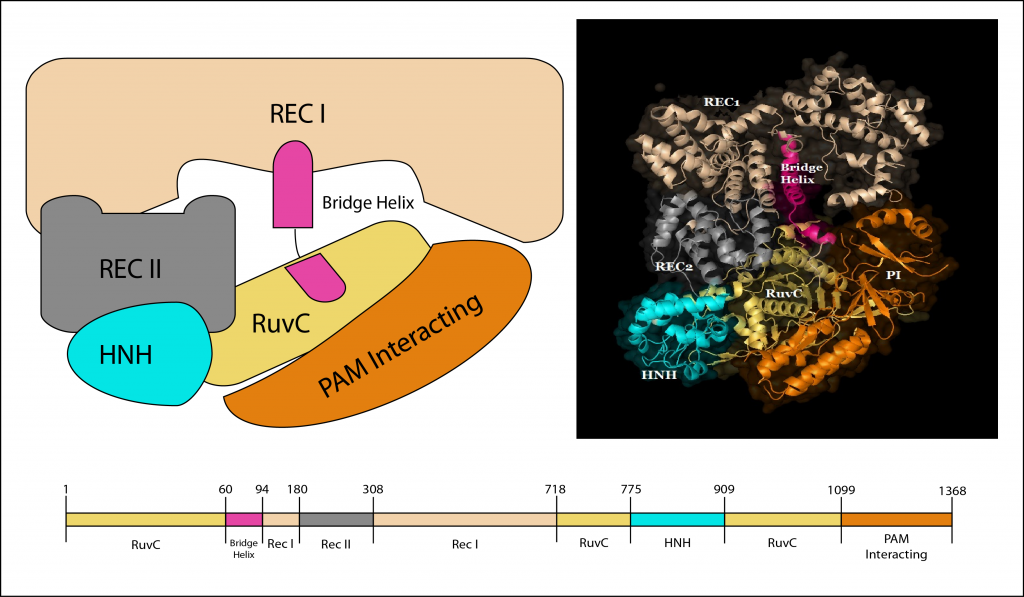
REC I là tiểu phần lớn nhất và có trách nhiệm định hướng cho gRNA. Vai trò của REC II vẫn chưa được hiểu rõ. Các cầu xoắn giàu arginine là rất quan trọng để bắt đầu hoạt động bám vào và cắt DNA mục tiêu (Nishimasu et al. 2014). Tiểu phần PAM-Interacting tạo cho PAM đặc tính chuyên biệt và do đó chịu trách nhiệm xúc tác cho sự bám của Cas9 vào DNA mục tiêu (Anders et al 2014;.. Jinek et al 2014; Nishimasu et al 2014;. Sternberg et al 2014.). Các tiểu phần HNH và RuvC là các nuclease, có vai trò cắt DNA sợi đơn. HNH và RuvC là những tiểu phần có độ tương đồng cao vì vậy chúng còn được tìm thấy ở những phân tử protein khác (Jinek et al. 2014; Nishimasu et al. 2014).
Các protein Cas9 sẽ ở trạng thái bất hoạt trong trường hợp có gRNA. Trong các hệ thống CRISPR/Cas9 nhân tạo, gRNA bao gồm một sợi đơn RNA tạo thành một hình chữ T (Hình 2). Các gRNA được thiết kế để có đầu 5’ là trình tự bổ sung với trình tự DNA mục tiêu.
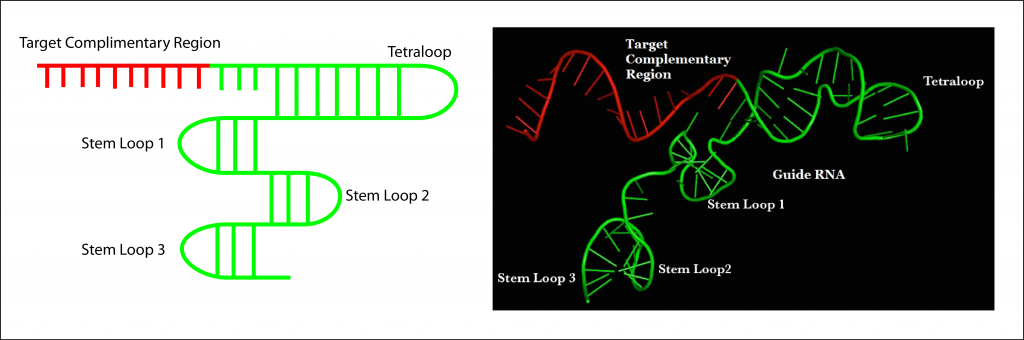
gRNA nhân tạo liên kết với các protein Cas9 và khi tương tác tạo thành phức hợp sẽ gây ra một sự thay đổi về hình dạng của protein (Hình 3). Sự thay đổi về hình dạng này sẽ kích hoạt Cas9 chuyển sang trạng thái hoạt động. Cơ chế của sự thay đổi về hình dạng không được hiểu hoàn toàn nhưng Jinek và các đồng nghiệp đưa ra giả thuyết rằng sự tương tác về không gian hay liên kết yếu giữa các tiểu phần protein và gRNA có thể là nguyên nhân gây ra sự thay đổi (Jinek et al. 2014).
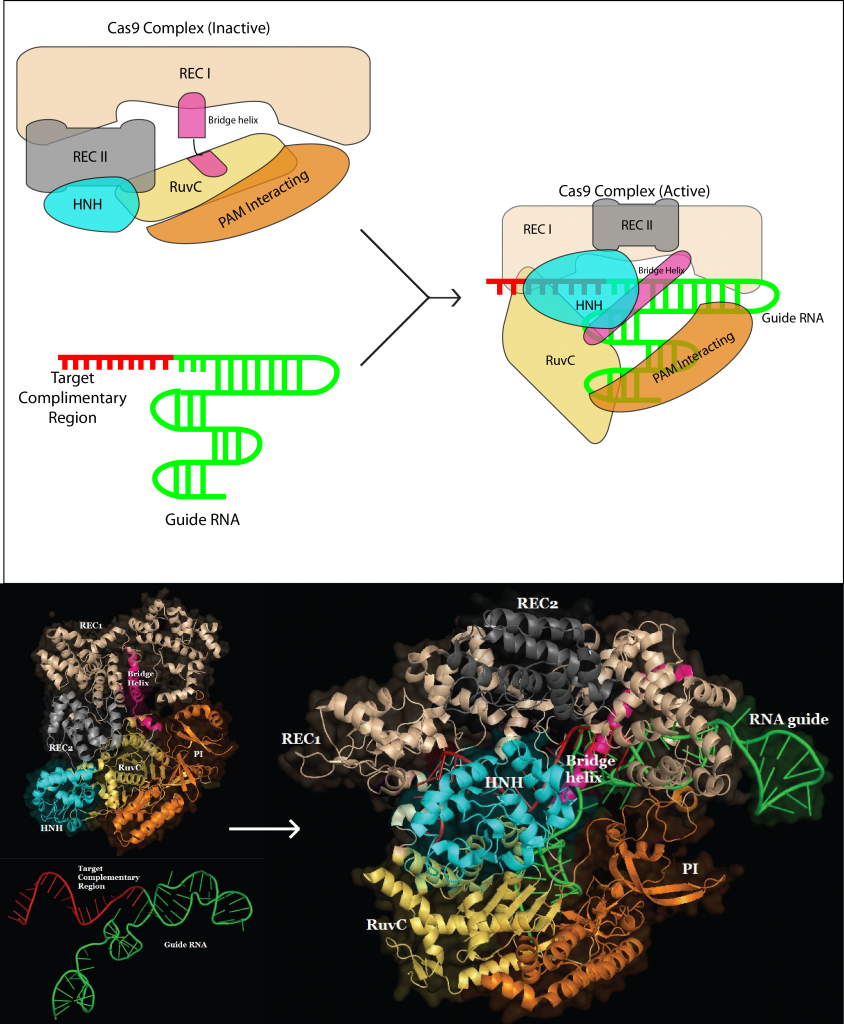
Khi các protein Cas9 được kích hoạt, nó sẽ tìm kiếm DNA mục tiêu bằng cách liên kết với các trình tự phù hợp với trình tự PAM (protospacer adjacent motif) của nó (Sternberg et al. 2014). Một PAM là một trình tự 2-3bp được gắn liền kề sau trình tự bổ sung của gRNA. Các trình tự PAM đã được xác định trong tất cả các hệ thống CRISPR, và các nucleotide cụ thể xác định PAM được chuyên biệt cho các loại hệ thống CRISPR (Mojica et al. 2009). PAM trong Streptococcus pyogenes là 5′-NGG-3 ‘(Jinek et al. 2012). Khi protein Cas9 tìm thấy một trình tự mục tiêu phù hợp với trình tự PAM, Cas9 sẽ tách các base phía trước trình tự PAM và bắt cặp với vùng bổ sung trong đoạn gRNA (Sternberg et al. 2014). Nếu vùng bổ sung và vùng mục tiêu bắt cặp đúng thì RuvC và HNH nuclease sẽ cắt DNA mục tiêu tại vị trí nuclotide thứ 4 phía trước PAM (Anders et al. 2014) tạo ra vùng DSB (double strand break) (Hình 4).
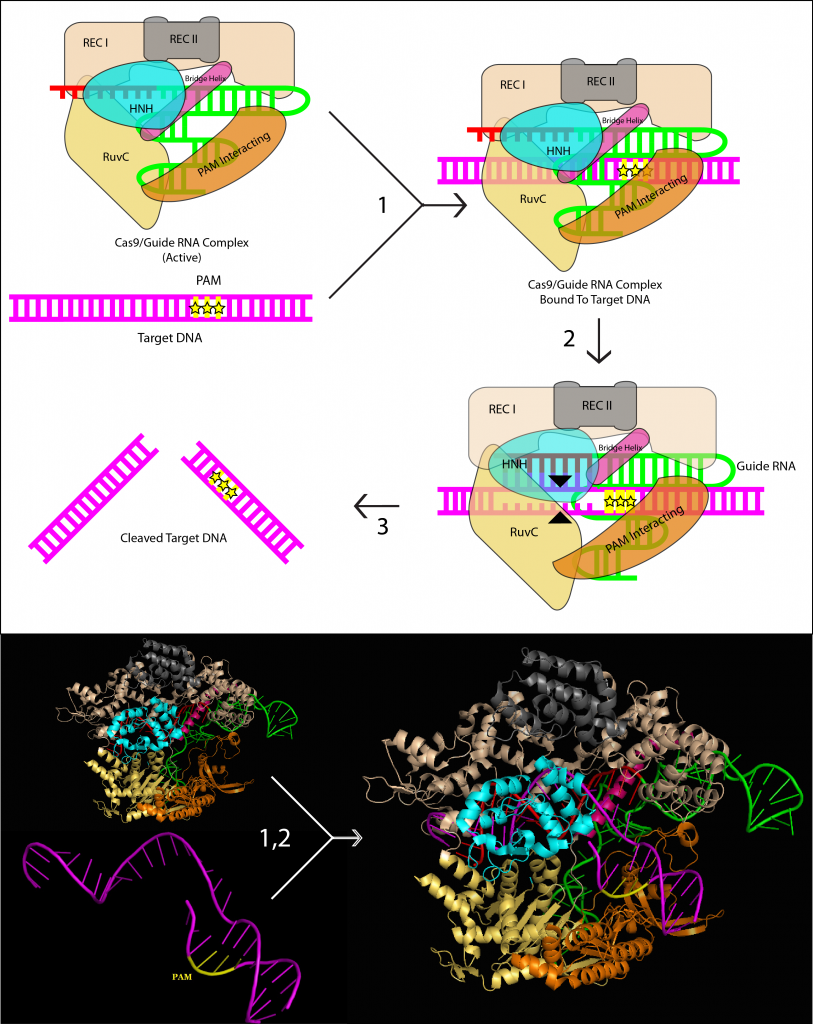
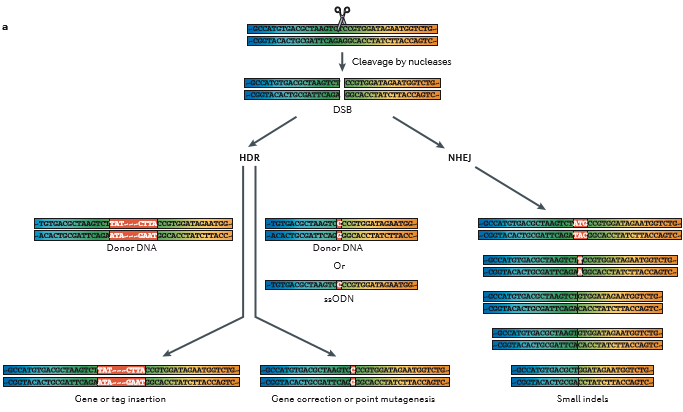
b | Khi hai DSBs được tạo ra trong cis trên một nhiễm sắc thể đơn bởi hệ thống enzyme nucleases, vùng ở giữa 2 vị trí cắt (flanking region) có thể bị loại bỏ hoặc ngược lại. c | Khi hai DSBs được tạo ra trên hai nhiễm sắc thể khác nhau, sự bắt cặp chéo (translocations) nhiễm sắc thể có thể được tạo ra
II: Ứng dụng linh hoạt CRIPSR/Cas9 trong chỉnh sửa bộ gen
1. Gen silencing/knockout
Cas9 endonuclease đã trở thành một công cụ phổ biến để chỉnh sửa gen trực tiếp trong hệ thống tế bào nhân thật (eukaruotic system). Với việc sử dụng một mục tiêu cụ thể CRISPR RNA (crRNA) và trans-kích hoạt crRNA (tracrRNA), hoặc một dạng hợp nhất gọi là RNA dẫn đường (gRNA). Sự phá vỡ bởi Cas9 tại những vị trí nhất định trong bộ gen phức tạp ở động vật có vú có thể được sửa chữa bởi các cơ chế sửa chữa DNA nội sinh thông qua một quá trình được gọi chung là sự bổ sung của những đoạn không tương đồng (NHEJ- non-homologous end-joining). Bởi vì NHEJ tạo ra những vị trí lỗi, xóa gen hoặc chèn (insertion/deletion-indels) có thể dẫn đến sự thay đổi khung cấu trúc gen và cảm ứng cơ chế kết thúc sớm để làm gen mục tiêu im lặng (gene silencing), tức không thể biểu hiện. Điều quan trọng cần lưu ý là sự chèn hoặc bị loại bỏ có kết quả từ NHEJ là ngẫu nhiên và khác nhau ở những tế bào khác nhau. Những thay đổi về gen một cách chính xác có thể được xác định bằng thực nghiệm bổ sung vào dòng tế bào vô tính. Các crRNA, tracrRNA, và gRNAs có thể được sao chép trong tế bào, trong ống nghiệm hoặc tổng hợp và biểu hiện thông qua sự biến nạp vào tế bào. Sự biểu hiện trong tế bào của Cas9 endonuclease có thể được thực hiện bằng plasmid hoặc vectơ biểu hiện lentivirus tích hợp điều khiển bởi các promoter hoặc cảm ứng.
2. DNA-free CRISPR-Cas9 gene editing
Hệ thống chỉnh sửa gen CRISPR/Cas9 với DNA-free có nghĩa là gì? Nó có nghĩa là hệ thống này không sử dụng các thành phần CRISPR-Cas9 dưới dạng vector DNA; mỗi thành phần là RNA hoặc protein. Bắt đầu với Cas9 mRNA hoặc protein Cas9 tinh khiết là nguồn cho Cas9 biểu hiện hoạt tính nuclease trong các thí nghiệm kỹ thuật gen có lợi thế cho một số ứng dụng. Tại sao? Việc sử dụng các Cas9 dựa trên DNA hoặc gRNA mang theo nó những khả năng biến đổi gen không mong muốn do plasmid DNA tích hợp vào các vùng cắt hoặc tích hợp vector lentivirus ngẫu nhiên. Vì lý do này, một hệ thống chỉnh sửa gen DNA-free có thể là một lựa chọn tốt cho việc tạo ra các dòng tế bào mong muốn. Nếu thử nghiệm liên quan đến việc quan sát của một kiểu hình trong quần thể mà không phải tế bào vô tính, các nhà nghiên cứu có thể không cần phải lựa chọn DNA-free. Tuy nhiên, nếu mục tiêu cuối cùng của thử nghiệm không yêu cầu làm giàu các tế bào Cas9 và tránh các sự tương tác có thể xem xét sử dụng Cas9 mRNA hoặc protein Cas9 tinh khiết.
3. Homology-directed repair (HDR)
Hệ thống CRISPR-Cas9 cắt DNA sợi đôi mục tiêu cũng có thể được sử dụng để tạo ra một gen knockin [4], chứ không phải là mục tiêu gen knockout. Sự chèn chính xác của một trình tự lạ có thể làm thay đổi vùng mã hóa của một gen để “sửa chữa” một đột biến, một loại protein, hoặc tạo ra một vùng giới hạn mới. Một vài nghiên cứu đã chứng minh rằng DNA sợi đơn có thể được sử dụng để tạo ra sự chèn chính xác bằng cách sử dụng crRNA và tracrRNA tổng hợp kết hợp với Cas9 nuclease. Ngoài ra, các hoạt động của Cas9 có thể được thay đổi để cắt chỉ một sợi thay vì sợi đôi. Các Cas9 nickase có thể được sử dụng với một cặp phức hợp crRNA: tracrRNA hoặc sgRNAs hướng mục tiêu đến hai vùng gần nhau trên 2 sợi đối diện, và khi được sử dụng với DNA sợi đôi ngắn, HDR có thể thực hiện.
4. Transient gene silencing or transcriptional repression (CRISPRi)
Với ứng dụng này, Cas9 được cải biến để nó không thể cắt DNA, và khi kết hợp với một gRNA nhằm vào vùng promoter mục tiêu, phức hợp này có thể làm giảm hoạt động phiên mã và biểu hiện gen đồng thời.
5. Transient activation of endogenous genes (CRISPRa or CRISPRon)
Bằng cách sử dụng một đột biến Cas9 mà nó không thể cắt DNA và một vùng kích hoạt phiên mã đã được hợp nhất, sự biểu hiện của gen nội sinh có thể tăng lên bởi protein dung hợp Cas9 hướng tới vùng promoter của gen đích nội sinh, hoặc nhiều gen đồng thời.
6. Embryonic stem cell and transgenic animals
Hệ thống CRISPR-Cas có thể được sử dụng như một kỹ thuật nhanh chóng và hiệu quả cho một hoặc nhiều thay đổi di truyền trên các tế bào gốc phôi của chuột nhằm tạo ra các thế hệ của những chuột con biến đổi gen. Một cách tiếp cận tương tự đã được sử dụng để biến đổi di truyền tế bào phôi đơn lẻ ở động vật linh trưởng.
7. Pooled genome-scale knockout screening
Các thư viện lentivirus đã được sử dụng MOI thấp để thực hiện sàng lọc hệ gen với khả năng tồn tại tế bào ung thư, pluripotency, và kháng thuốc. Với việc tích hợp số lượng bản sao duy nhất, bi-allelic gene silencing có thể được thực hiện với tần số cao. Đại diện tương đối của các gen tham gia vào tạo ra các kiểu hình quan trọng của quá trình sàng lọc (ví dụ tế bào chết, tăng sinh, kháng thuốc, vv) có thể được xác định bởi trình tự của các thế hệ tiếp theo
III: Phương thức sử dụng CRISPR/Cas9 tạo đột biến indel knockout gen mục tiêu

NHEJ được sử dụng như là một cơ chế sửa chữa tích cực nhất nhưng thường dẫn đến indels gần trình tự mục tiêu. Nếu indel xảy ra trong khung đọc ORF (open reading frame), nó có thể làm thay đổi khung ORF dẫn đến tín hiệu của mã kết thúc sớm (premature stop codon) qua đó loại bỏ các chức năng ban đầu của gen-tắt gen gây bệnh. Các phân tích gen như SURVEYOR hoặc giải trình tự gen (Sanger hoặc NGS)[8] sau đó sẽ giúp xác định các tế bào có chứa indels ở gen mục tiêu hay không. Ngoài ra, bằng cách sử dụng cơ chế sửa chữa không hoàn hảo của tế bào, CRISPR cho phép các nhà khoa học chỉ đơn giản là xây dựng thư viện các dòng tế bào với knockout indel tại những vùng của bộ gen muốn nghiên cứu.
NHỮNG BƯỚC CẦN THIẾT KHI BẮT ĐẦU THAO TÁC THÍ NGHIỆM VỚI CRISPR/CAS9 TRÊN TẾ BÀO:
Bước 1: Xác định loại tế bào sử dụng và trình tự gen mục tiêu
Bước 2: Thiết kế các trình tự oligo và primer cần thiết: gRNA, Fw, Rv
Sử dụng công cụ CRISPR Design Tool: http://tools.genome-engineering.org
Hoặc Benchling: https://benchling.com/academic
Lựa chọn trình tự mục tiêu (ví dụ, một đoạn gen 1-kb từ các khu vực quan tâm), xác định và phân loại các vùng mục tiêu phù hợp. Các thông số được thiết lập giúp tính toán cho từng mục tiêu dự định. Ngoài ra, công cụ còn có thể chọn trình tự gRNA bằng cách xác định trình tự 20 bp bất kỳ với đầu 5¢-NGG.
Bước 3: Tổng hợp gRNA [7]
Có thể tổng hợp gRNA bằng phương pháp PCR hoặc phương pháp cloning
Việc lựa chon phương pháp nào sẽ phụ thuộc vào dòng tế bào lựa chọn để thao tác và mức độ hiệu quả vận chuyển đến gen mục tiêu
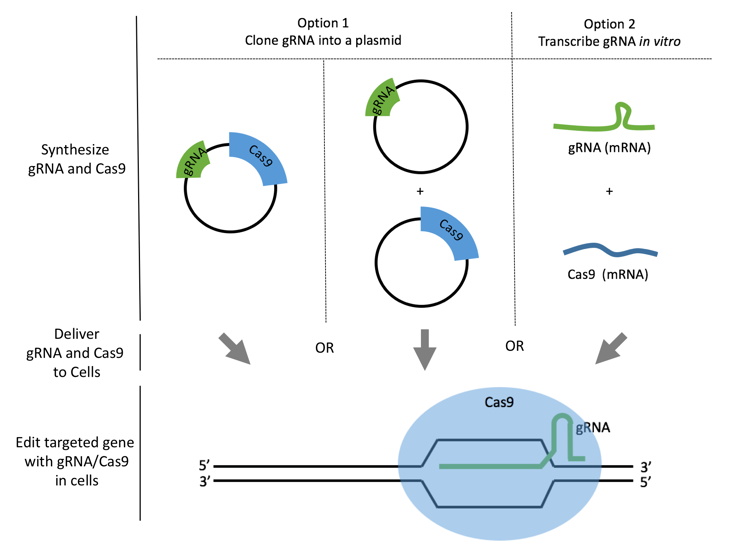
Clone gRNA vào plasmid
Quy trình tham khảo từ phòng lab của giáo sư Feng Zhang tại Viện MIT: clone gRNA vào plasmid PX330
Protocol:
- Bước 1: Order oligo cho việc tổng hợp gRNA
- Bước 2: Phosphorylate and anneal oligo
- Bước 3: Cắt vector với enzyme cắt giới hạn (restriction enzyme-RE) BbsI và nối với oligo gRNA
- Bước 4: Biến nạp plasmid vừa nối ở bước 3 vào tế bào
- Bước 5: Chọn những khuẩn lạc và phân tích xác định trình tự bằng phương pháp giải trình tự
Ưu điểm và nhược điểm của phương pháp:
Ưu điểm:
- Sau khi được clone vào plasmid, gRNA sẽ dễ dàng xâm nhiễm vào tế bào như HEK (human embryonic kidney) với cấu trúc vòng siêu xoắn (supercoiled) của plasmid
- Plasmid dễ dàng sao chép và có thể dự trữ lâu dài cho các quy trình nghiên cứu lớn.
Nhược điểm:
- Dạng plasmid sẽ dẫn đến việc xâm nhiễm vào tế bào của gRNA gặp khó khăn đối với một vài dạng tế bào như tế bào gốc phôi (human embryonic stem cell)
Tổng hợp gRNA bằng phương pháp PCR
Phương pháp này cần bổ sung trình tự T7 promoter vào trình tự gRNA trong quá trình thiết kế oligo gRNA. Phản ứng PCR được thực hiện như một phản ứng tổng hợp mRNA và sử dụng enzyme T7 polymerase cho quá trình tổng hợp
Protocol tham khảo từ phòng lab của giáo sư Jennifer Doudna
- Bước 1: Order oligo cho việc tổng hợp gRNA
- Bước 2: Tổng hợp gRNA như một mRNA bằng phương pháp PCR
- Bước 3: Tinh sạch và xác định hàm lượng mRNA
Ưu điểm:
- Là lựa chọn ưu việt cho việc xâm nhiễm gRNA vào các tế bào khó xâm nhiễm như tế bào gốc phôi
- RNA chỉ tồn tại trong thời gian ngắn. Vì vậy, hện thống gRNA và Cas9 xâm nhiễm tế bào dạng mRNA sẽ bị phân hủy nhanh chóng sau đó nên sẽ không di truyền cho các thế hệ tế bào tiếp theo.
Nhược điểm:
- Không có phương pháp cho việc sàng lọc các tế bào có chứa hệ thống gRNA và Cas9
- Cần kinh nghiệm thao tác trên RNA để đảm bảo việc tổng hợp mRNA đạt hiệu quả cao.
Bước 4: Vận chuyển phức hợp gRNA-Cas9 đến tế bào mục tiêu[9]
Việc vận chuyển phức hợp gRNA/Cas9 đến tế bào mục tiêu có thể theo 3 phương pháp khác nhau: Lipofection, Electroporation, Lentiviral transduction
Việc lựa chọn phương pháp nào sẽ phụ thuộc vào dòng tế bào lựa cũng như những ưu và nhược điểm của mỗi phương pháp.
Lipofection

Lipofection là phương pháp dựa trên đặc tính của liposome mang điện tích dương sẽ liên kết với DNA/RNA mang điện tích âm tạo nên một phức hợp mang mạng lưới điện tích dương từ đó giúp việc tiếp cận (bám) và xâm nhập vào tế bào một cách dễ dàng hơn
Quy trình thực hiện khá đơn giản: Chuẩn bị hỗn hợp chứa gRNA/Cas9 và tiểu bào lipid (lipid visicles) để tạo thành phức hợp liposome-gRNA/Cas9. Sau đó, cho trực tiếp phức hợp này vào môi trường chứa tế bào. Nuôi ủ 24h và tế bào bắt đầu biểu hiện gRNA/Cas9
Ưu điểm:
- Quy trình đơn giản, dễ thao tác và không yêu cầu bất kỳ thiết bị phức tạp nào
- Linh động vì có thể vận chuyển cả DNA hoặc RNA vào tế bào
Nhược điểm:
- Không là phương pháp được lựa chọn cho việc vận chuyển cho nhiều loại tế bào, chẳng hạn tế bào gốc phôi
Electroporation
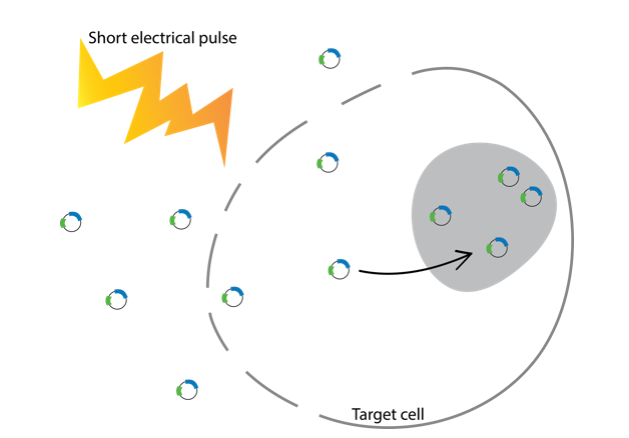
Electroporation là phương pháp sử dụng dòng điện 300-400mV để tạo ra xung động đến tế bào trong khoảng thời gian ít hơn 1/1000 giây. Xung động này giúp tạo ra những lỗ tạm thời trân màng tế bào và thông qua đó gRNA/Cas9 xuyên qua để đi vào tế bào đến locus mục tiêu.
Ưu điểm:
- Nhanh, tốn ít thời gian. Vì vậy, có thể vận chuyển gRNA/Cas9 vào một số lượng lớn tế bào trong thơi gian ngắn
Nhược điểm:
- Cần thiết bị chuyên biệt cho phương pháp này
- Cần khảo sát thời gian và cường độ dòng điện phù hợp để tránh sự chết của tế bào trong suốt quá trình thực hiện.
Lentiviral transduction

Lentiviral transduction là phương pháp sử dụng lentiviral vector để vận chuyển gRNA/Cas9 vào tế bào thông qua cơ chế xâm nhiễm của virus. Lentiviral vector có nguồn gốc từ một lớp của Retrovirus-gây ra suy giảm hệ thống miễn dịch ở người (HIV-1)
Quy trình thực hiện:
Plasmid chứa gRNA/Cas9 trước hết cần được đưa vào tế bào 293T để tạo ra các hạt lentivirus chức năng mang gRNA/Cas9. Các hạt này được thu nhận và cho tải nạp (transduction) vào tế bào mục tiêu. Sau khi vào tế bào, RNA của virus bao gồm cả gRNA/Cas9 sẽ phiên mã ngược, đi vào nhân và tiến đến các loci một cách ngẫu nhiên.
Ưu điểm:
- Hiệu quả xâm nhiễm/tải nạp cao kể cả những dòng tế bào khó như tế bào thần kinh
Nhược điểm:
- Quy trình thực hiện phức tạp, mất nhiều thời gian. Mất ít nhất 2 tuần để tải nạp các hạt lentiviral vào tế bào mục tiêu.
- Không thể kiểm soát vị trí tương tác giữa bộ gen virus và gen tế bào mục tiêu. Một vài trường hợp có thể ảnh hưởng đến chức năng tế bào. Vấn đề này có thể được xem xét và thử với adeno-associated virus (AAV) và sẽ cần thêm thời gian cho việc này.
Bước 5: Phát hiện các indel
Có 2 cách để phát hiện các indel bao gồm SURVEYOR và giải trình tự [8]
Bước 6: Xác định hiệu quả knockout gen hay hiệu quả chỉnh sử gen mục tiêu của hệ thống CRISPR/Case
- Phân tích bằng RFLP
- Phân tích bằng phương pháp giải trình tự: Sanger, NGS [8]
IV: Ưu điểm và hạn chế của hệ thống CRISPR/Cas9 so với các kỹ thuật sửa đổi gen khác [8]
1. Ưu điểm của hệ thống CRISPR/Cas9 [10]
- Thao tác linh động: Cas9 có thể dễ dàng retargeted các trình tự DNA mới bằng cách thiết kế những cặp oligos khác mã hóa các gRNA tại những mục tiêu khác. Ngược lại, retargeted của Talen đến một trình tự DNA mới đòi hỏi việc thiết lập hai gen Talen mới và điều này sẽ làm mất nhiều thơi gian hơn.
- Cắt chuyên biệt với độ đặc hiệu cao: WT S. pyogenes Cas9 (SpCas9) được xác định cắt theo dạng đầu bằng (blunt end) tại vị trí base thứ 17 và 18 trong trình tự DNA mục tiêu. Đột biến dư lượng xúc tác trong RuvC hoặc HNH nuclease của SpCas9 biến đổi enzyme thành enzyme DNA nicking. Ngược lại, Talens tách không đặc hiệu trong mối liên kết 12-24 bp giữa những cặp base của vùng Talen monomer-binding.
- Chỉnh sửa gen hiệu quả: SpCas9 và Talens đều được biết như những kỹ thuật chỉnh sửa gen hiệu quả trong nhiều loại tế bào và sinh vật. Tuy nhiên, do sự dễ dàng nhắm mục tiêu, Cas9 có thể được sử dụng để nhắm mục tiêu nhiều locus gen đồng thời bởi sự kết hợp của gRNAs đến các tế bào quan tâm.
2. Hạn chế của hệ thống CRISPR/Cas9 [10]
- Nhắm sai mục tiêu bởi sự lựa chọn sai mục tiêu ban đầu
- Sự thay đổi tương đồng chỉ trên một số rất ít tế bào
- Tỷ lệ không hiệu quả của HDR và NHEJ
- Hiệu quả vận chuyển đến tế bào và các thành phần khác
- Sự an toàn và hiệu quả
- Vấn đề đạo đức về những cạnh tranh phi khoa học như chính trị, thương mại, hay chiến tranh.
- Sự hiểu biết đối với toàn bộ gen con người còn nhiều hạn chế, nhất là sự biểu hiện của gen: những gen nào thực sự là bản sao của chính nó và những gen nào chỉ là sự gây nhiễu về tín hiệu? Những hậu quả tiềm tàng trong việc chỉnh sử gen, thiếu tính cụ thể trong mục tiêu, nhắm mục tiêu không bao hàm đầy đủ, tất cả đều có thể có tác động tiêu cực đối với tế bào sau chỉnh sửa gen.
Tham khảo:
[1] https://sites.tufts.edu/crispr/crispr-mechanism/
[2] CRISPR-Cas9 Genome Editing, Nature Collection, S11 (2016)[3] http://dharmacon.gelifesciences.com/applications/gene-editing/
[4] https://en.wikipedia.org/wiki/Gene_knockin
[5] https://blog.benchling.com/how-to-create-knockouts-using-crispr/
[6] https://www.addgene.org/crispr/guide/
[7] https://blog.benchling.com/how-to-synthesize-your-grnas-for-crispr/
[8] Ran, F.A., et al., Genome engineering using the CRISPR-Cas9 system,doi:10.1038/nprot.2013.143
[9] https://blog.benchling.com/how-to-express-crispr-in-your-target-cells/
[10] George M.Church., et al., CRISPR-Cas9 System: Opportunity and Concern, doi:10.1373/clinchem.2016.263186
Nguồn hình minh họa: http://www.nature.com/nrc/journal/v15/n7/full/nrc3950.html
Tác giả: IBSG Molecular Cell Biology
Cố vấn và biên tập: Tiến sĩ Nguyễn Ngọc Phương Thảo





